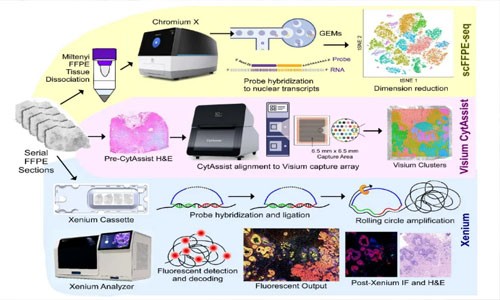
解决方案
仪器平台
伯豪生物服务科技创新,平均2天协助客户发表一篇SCI文章!
关于上海伯豪生物技术有限公司
上海伯豪生物技术有限公司(简称:伯豪生物,英文名:Shanghai Biotechnology Corporation)2008 年 12 月成立,是生物芯片国家工程研究中心的有机组成部分,专注于提供专业的生物医药和疾病诊断创新技术、产品和优质服务。伯豪生物自成立以来获得了国家基因检测技术应用示范中心、国家工信部专精特新“小巨人”企业、院士专家工作站、高新技术企业、上海市研发公共服务平台(负责“上海市高通量疾病标志物服务平台”和“上海市基因芯片专业服务平台”的运营)、上海市服务外包公共平台、上海科技小巨人企业、浦东新区企业研发机构、联合实验室建设等一系列资质,拥有医疗机构执业许可证、医疗器械经营许可证。
了解更多+
服务科技创新,护航人类健康!www.shbio.com
29
2024-03
-
2024-03-22
-
2024-03-21
-
2024-03-20
合作伙伴
<